シークエンサーの仕組みと「1,000ドルゲノム」時代
公開日:2016年08月19日
ヒトの全ゲノムの解読が約13年の歳月と約3500億円の費用をかけて完了したのが2003年のこと。それが今では、約10万円の費用をかければ1日足らずで済むほど、ゲノム解析の技術革新が進んでいます。

飛躍的に高速、安価になったゲノム解読
ヒトのゲノムの全塩基配列を解析するプロジェクト、ヒトゲノム計画がスタートしたのは、1990年。その後、塩基配列のドラフト版完成の発表が2000年4月に行われ、完成版の公開が2003年4月になされました。この間に費やされた費用は約3500億円にのぼったそうです。しかし、その後のゲノムの解読装置(シークエンサー)の発展は目覚ましく、今では10万円前後でヒト1人分のゲノムが解読できるほどになっています。解読スピードも飛躍的に向上し、ヒトゲノム計画で主力として活躍したシークエンサー「ABI3700」(ABI社)が、1回のランで17,000個の塩基配列を決定できたのに対して、2014年1月に発表された次世代シークエンサーの「HiSeq2500」(イルミナ社)では、1回のランで1.8兆個の塩基配列を決定でき、3日間で16人の全ゲノム配列を30x(各塩基を平均30回読む)の高い信頼性で決定することが可能になっています。
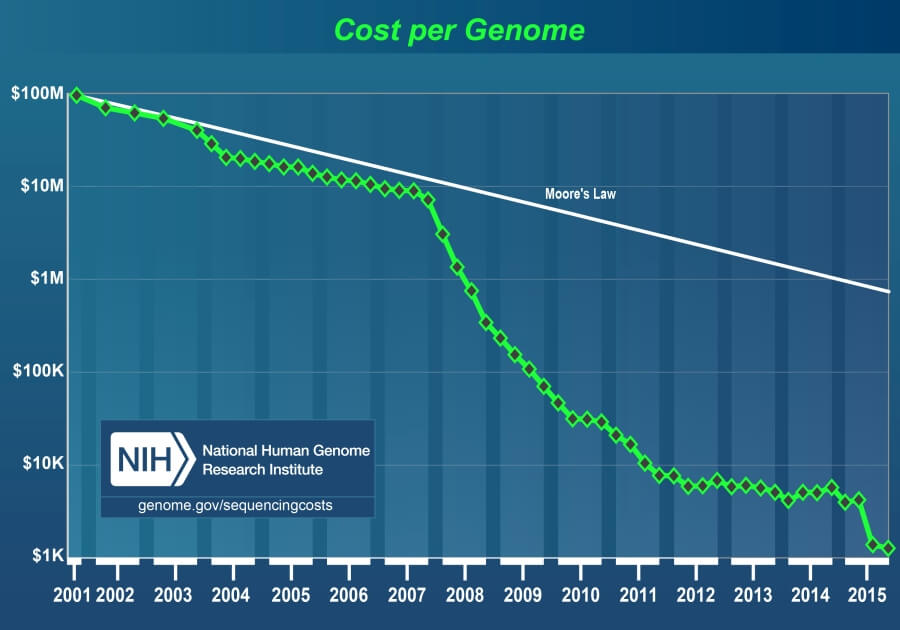
ヒト1人分のゲノム解読にかかる費用の推移
NIH Natinonal Human Genome Research Institute より
ゲノム解読の仕組み
ゲノム解読とは、塩基の種類(アデニン(A)、シトシン(C)、G(グアニン)、T(チミン))と順番(塩基配列(シークエンス))を調べることです。しかしDNAは小さいので、直接観察して塩基配列を調べることは困難です。そこで解読するDNAをPCRなどで増幅させた後、まるでパズルのような作業を行うのですが、ここではそのようなのシークエンサーの仕組みを紹介したいと思います。
第一世代シークエンサーの解読方法

ABI3700(ABI社)
第一世代のシークエンサーでは、イギリスのフレデリック・サンガー博士が発明した鎖停止法(別名:サンガー法)が用いられています。ちなみにフレデリック・サンガー博士はこのDNAの塩基配列決定とインスリンの構造研究によって、ノーベル化学賞を2度受賞、これまでにノーベル化学賞を2度受賞したのは、このサンガー博士だけです。
1、増幅させた2本鎖DNAから1本鎖DNAへ
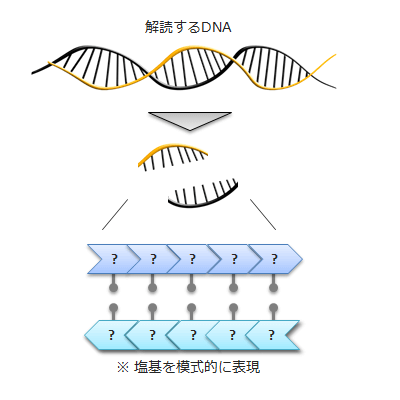
増幅させたDNAを2本鎖から1本鎖の状態へ分解。50~400塩基ごとに人工的にぶつ切りにされる。
2、塩基と反応
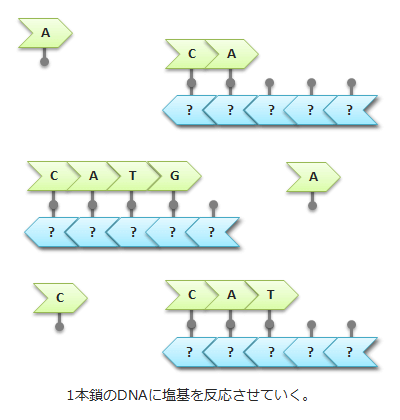
DNAの片側の鎖と多数の塩基を反応させると、アデニン(A)はT(チミン)と、シトシン(C)はG(グアニン)と自動的にくっついていく。ただし、ヒトゲノム計画が終了した2003年ごろには、まだ、この塩基の直接検出する方法がなかった。
3、蛍光分子付きの塩基
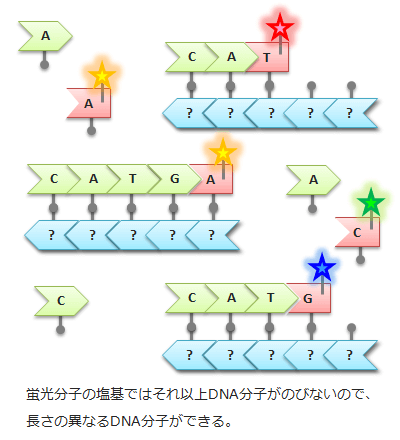
そこで、反応させる塩基の中に、人工的に蛍光分子をくっつけ、さらに塩基のつながりがそれ以上伸びないように細工された塩基を混ぜる。
塩基が自動的につながっていく際、人工的な塩基がどこに入ってくるかはランダムなので、DNAの端に蛍光分子がくっついた、1塩基、2塩基、3塩基…の長さのDNAが作られる。
4、電気泳動
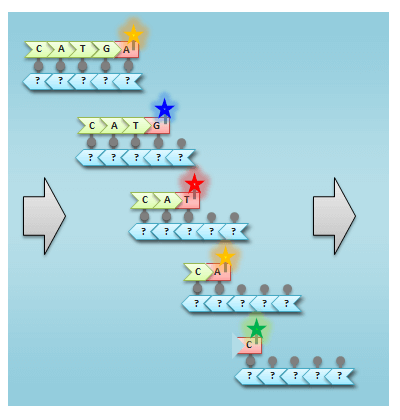
これらのDNAを電気泳動装置などの中に流すと、長さの短いDNAほど早く流れる。
5、蛍光の検出
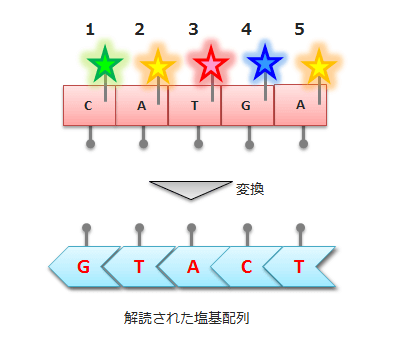
装置を通過する順に蛍光を検出すれば、蛍光分子のついた塩基の種類と位置が分かる。
ただし、検出した塩基は、調べたいDNAとA⇔T、C⇔Gが逆なので、それらを反転させてはじめて調べたいDNAの塩基配列が分かる。
次世代シークエンサーの解読方法
2005年ごろから次世代シークエンサーと呼ばれる高速解読装置が各社から次々と発表されるようになりました。
従来の方法と大きく異なるのは、並列処理できるDNA断片の数です。イルミナ社の資料 によると、従来の方法が、1~96のDNA断片を同時処理していたのに対して、次世代シークエンサーは、数千万から数億のDNA断片を処理するそうです。これによって、解析スピードが飛躍的に向上しました。
1、増幅させた2本鎖DNAから1本鎖DNAへ

解読するDNAを増幅させるのは、従来型の方法と同じ。
2、ピロリン酸、水素イオンまたは蛍光の検出
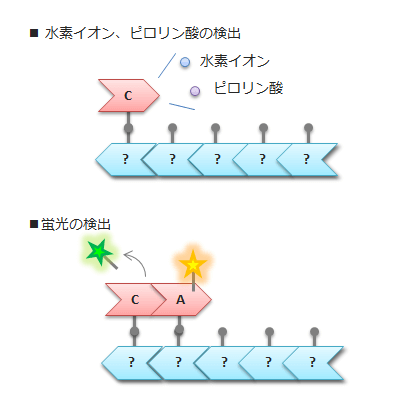
従来型とは異なり、先につなげる塩基を1つずつ1本鎖DNAと反応させていく。以下の2つの方法が現在主流。
- 通常の塩基がくっつくときだけ放出されるピロリン酸、水素イオンを検出するタイプ(Ion Proton(ライフテクノロジーズ社) など)。
- 従来型同様蛍光を検出するタイプ。従来型との違いは、検出した蛍光分子は次の塩基がつながるときには外れるので、蛍光が混ざることがないので、連続して検出することが可能(HiSeq 2500(イルミナ社) など)。
ペアになる塩基を反応させたときだけ塩基がくっつき、水素イオン、ピロリン酸または蛍光が発せられるので、それを検出すれば、塩基の種類が分かる。
3、塩基反応の繰り返し
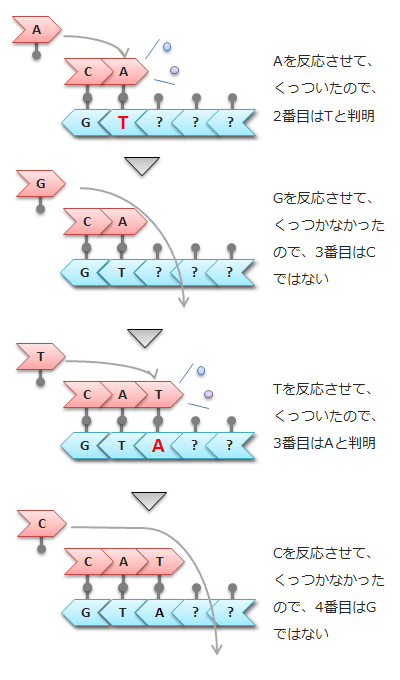
4種類の塩基を順番に反応させ、それを繰り返すことで、順次塩基配列が特定されていく。従来型のように、長さの異なるDNAを一通り用意する必要がないため、効率的。
第三世代シークエンサーの登場

PacBio RS II(パシフィックバイオサイエンス社)
さらに1分子配列決定技術と呼ばれる、PCRによるDNAの増幅作業や塩基配列決定のための反応処理が必要ない第三世代のシークエンサーの研究・開発がすすめられています。中にはアメリカのパシフィックバイオサイエンス社の「PacBio RS II」のようにすでに実用化されているものもあります。
国内では、大阪大学の川合知二教授が開発した、量子力学に基づく1分子計測技術「ゲーティングナノポア」と呼ばれる技術が注目されていて、今後の実用化が期待されています。
Scientific Reports 2, Article number: 501 (2012)
Single-Molecule Electrical Random Resequencing of DNA and RNA
このページが役に立ったらシェアお願いします!
あわせてよく読まれる記事
-
遺伝子とは?DNAとは?
DNAとはデオキシリボ核酸と呼ばれる「物質」、遺伝子とはそのDNAの上に書かれたタンパク質の設計図、つまり「情報」です。遺伝子に変化が生じてタンパク質の設計図が変わってしまうと、そのタンパク質が担っていた機能がうまく働かなくなる場合があります。…
-
DNAチップを使ったSNPの型の調査方法
遺伝子検査におけるSNPの調査には、DNAチップが主に使われます。このページでは、このDNAチップを使って、SNPの型をどのように調べるかについて、紹介します。…
-
乳癌(乳がん)の遺伝子検査を受ける前に知っておきたいこと
乳癌(乳がん)の遺伝子検査について、検査対象となるBRCA1、BRCA2遺伝子のこと、遺伝子検査の流れと費用、病院での検査とネットなどから申し込める検査との違いなどをまとめました。…